郎曌博研究组开发出高效的双子叶植物腺嘌呤单碱基编辑器
2021年10月25日,Plant Biotechnology Journal 在线发表了中国科学院分子植物科学卓越创新中心郎曌博研究组题为“Efficient A·T to G·C Base Conversions in Dicots Using Adenine Base Editors Expressed under the Tomato EF1α Promoter”的研究论文。该研究工作中,研究者为了提高腺嘌呤单碱基编辑器在双子叶植物中的编辑效率,从7种不同类型的启动子驱动的腺嘌呤编辑器中筛选出由番茄pSlEF1α启动子驱动ABEs效率最高,番茄中最高可达71.4% ,大豆根毛的编辑效率可达50%以上。此外,研究者还拓展了pSlEF1α驱动的单碱基编辑器的Cas9 变体系统,在拟南芥,番茄,大豆等双子叶植物的NG PAM部分位点也可实现精准编辑。因此,该系统大大提高的腺嘌呤单碱基编辑器在双子叶植物的编辑效率和编辑范围,为科研和育种提供强有力的工具,为基因组编辑种质创新提供了新思路。
植物的许多重要农艺性状与单核苷酸多态性有关。Ubiquitin启动子驱动的ABE在水稻和小麦中表现良好。然而,双子叶植物中ABEs的低效率仍然是一个重大问题。先前在双子叶拟南芥中的一项研究表明,35S和Yao启动子驱动的ABE几乎没有活性,但RPS5A启动子驱动的ABE成功地在拟南芥中引起A-to-G转化,这表明ABE系统中使用的启动子可以显著影响编辑效率(Kang等人,2018)。这表明可以通过优化编辑系统的启动子元件是提高ABE的效率一种手段。
为了筛选在番茄中驱动ABEs的有效启动子,研究者根据已发表的RNA表达数据,选出7种不同的启动子在番茄(Ailsa Craig)中引导ABEs的表达,包括35S、AtUbi、ATRP5A、SlRPS5A1、SlRPS5A2、SlTCTP和SlEF1α启动子(图1a)。通过比较了7个ABE单碱基编辑系统在6个靶位点的编辑效率检测,我们发现p35S-和pAtUbi-驱动的ABEs在番茄中所有测试的sgRNA位点几乎没有活性(图1b)。pSlEF1α-ABE-nCas9在不同的靶位点表现出具有非常高的编辑效率。此外,论文中统计了该系统的编辑窗口,为guide RNA上的第4-11位的腺嘌呤。同时论文中也检测了脱靶效率,在所编辑的位点未发现脱靶。
研究者在大豆毛状根中进一步确认了ABEs编辑系统的有效性。在sgRNA19和sgRNA20上,pSlEF1α驱动的ABE比p35S或pSlRPS5A1驱动的ABE具有更高的编辑效率(图1g)。这些结果表明,pSlEF1α-ABEs系统在双子叶作物(如番茄和大豆)中都具有高效的A-to-G的效率。同样为了拓展该系统在在基因组中的靶位点选择,作者利用pSlEF1α引导的ABE7.10-nCas9-NG和TadA8e-nCas9-NG在番茄和大豆的一些NG PAM靶位点上成功实现了A-to-G的编辑。这些结果表明,SlEF1α启动子可用于驱动spCas9变异体介导的ABE在一些NG PAM位点同样能进行有效的腺嘌呤碱基编辑。
综上所述,基于我们的研究筛选出的pSlEF1α启动子显著提高ABEs系统在双子叶植物中的单碱基编辑效率,为提高双子叶植物编辑效率提供了新思路,此外,该启动子在不同系统中显示出优良的适用性,扩展了在NG PAM位点的编辑范围,丰富了植物基因编辑系统类型,也为作物品种改良等提供了更广泛、高效、精准的新工具,为基因组编辑种质创新提供了新思路。
中国科学院分子植物科学卓越创新中心的郎曌博研究员为论文的通讯作者,牛庆丰博士、博士研究生吴思群和山东舜丰基因编辑研究院的谢洪涛博士为本文的共同第一作者。该工作得到了中国科学院先导项目、国家自然科学基金青年科学基金项目等经费的资助。
论文链接:https://onlinelibrary.wiley.com/doi/abs/10.1111/pbi.13736
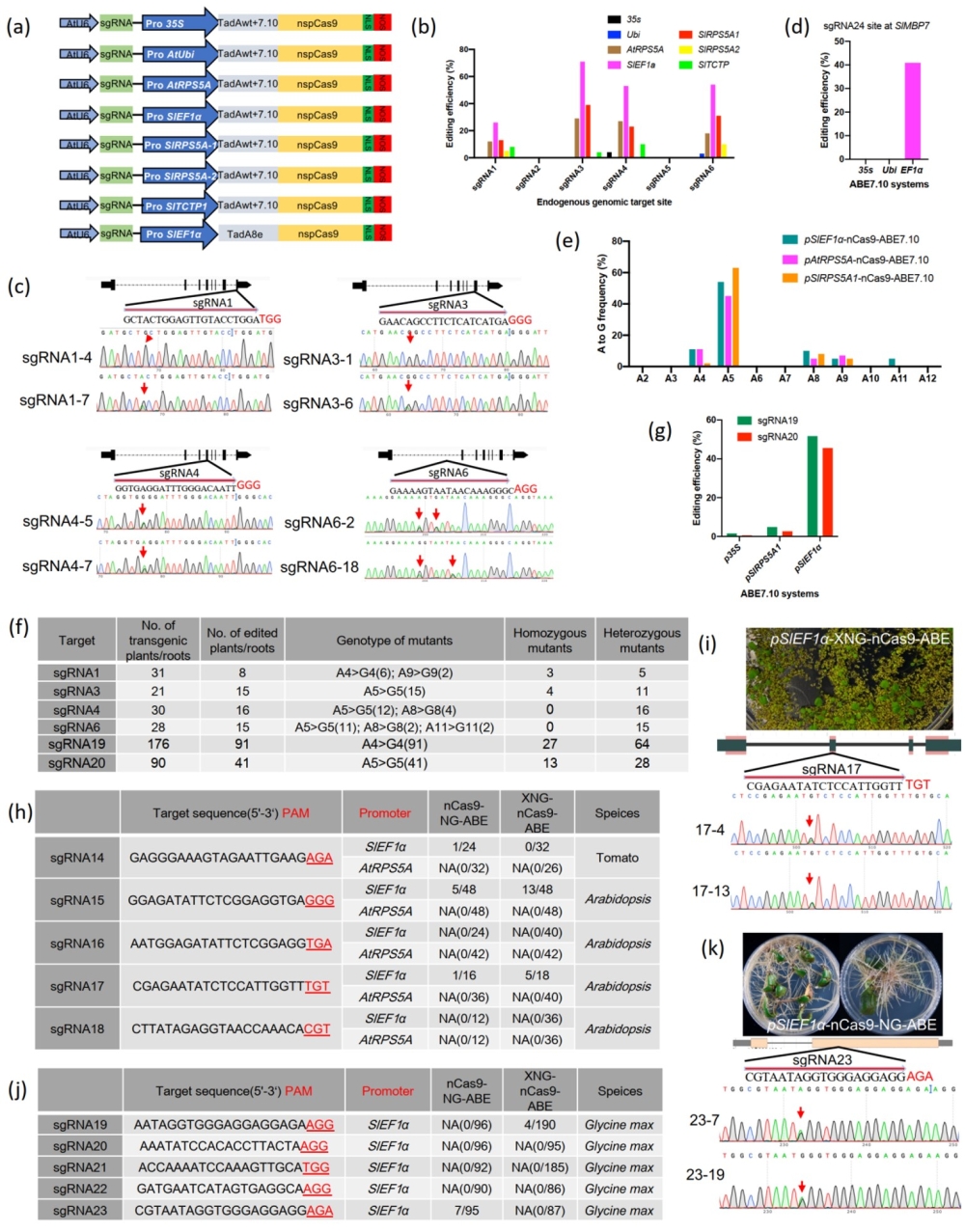
Figure 1. Analyses of adenine base editing efficiencies.