徐永镇研究组在反式剪接的发生机制研究中取得重要进展
2015年4月1日《Genes & Development》发表了植物生理生态研究所徐永镇研究组题为“A conserved intronic U1 snRNP-binding sequence promotes trans-splicing in Drosophila”的研究论文。
RNA剪接通常发生在一个前体RNA (pre-RNA) 的分子内部,即‘顺式剪接’(cis-splicing),但也可以发生在两个前体RNA分子之间,最终生成杂合的mRNA,称为‘反式剪接’(trans-splicing)。发生反式剪接的两个前体RNA分子可以来自同一基因的同义链或正反义链,甚至来自于不同染色体上的两个基因。已知的反式剪接基因数量相对较少,但普遍存在于真核生物中。低等真核生物,如锥虫与线虫,其绝大多数基因都需要进行反式剪接的加工,上世纪90年代的研究证明它们是由SL (Spliced Leader) RNA促进发生的。然而,高等真核生物体内并没有发现SL RNA存在,其反式剪接发生机制一直都不甚清楚。为了研究这个科学问题,徐永镇研究组以家蚕和果蝇为材料,系统地开展了昆虫的反式剪接研究。
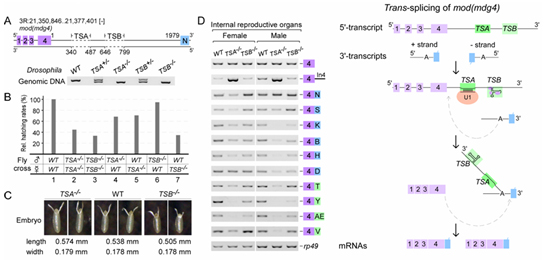
前期通过转录组测序与实验验证,确认了家蚕中存在20个基因的65个反式剪接事件,发现其中的一些反式剪接产物阅读框可以编码完整的、来自于两个基因的蛋白质结构域,这在基因进化研究上具有重要的理论意义(Shao et al., 2012, RNA)。mod(mdg4)和lola是昆虫中最经典的两个反式剪接基因,它们分别与昆虫的染色质重建和神经元功能密切相关。徐永镇课题组以mod(mdg4)为研究对象,通过细胞水平上的多种截短体和突变体研究,发现该基因反式剪接的发生取决于其5’共同转录产物的最后一个内含子中两个RNA片段,TSA与TSB。TSA与TSB的核心序列在基因组序列已知的12个果蝇属昆虫中高度保守。采用CRISPR/Cas9技术,在黑腹果蝇基因组中特异性地分别敲除这两个RNA片段,导致胚胎发育异常,孵化率明显下降。重要的是研究进一步证明TSA元件为mod(mdg4)基因反式剪接所必需,通过碱基配对,其核心的13 nt序列能够特异性地结合U1 snRNP,促使了反式剪接的发生, 并且TSA元件的单独存在就足以诱导反式剪接的发生。TSB元件具有十分保守的二级结构,起到反式剪接发生的增强子作用。研究还显示TSA元件也存在于昆虫的其它一些反式剪接基因中,如lola,从而揭示出高等真核生物中存在的一种新的反式剪接发生机制。
该研究工作主要由博士生高俊丽、副研究员樊玉杰、博士生王修业在徐永镇研究员指导下共同完成,得到了国家自然基金委、科技部和中科院项目的资助。